서 론
명월초(Gynura procumbens)는 국화과의 여러해살이 초본으로 중국, 말레이시아, 인도네시아, 베트남 등에서 전통적인 약초로 사용되는 식물이다(Kanzil and Pritesh, 2016). 최근 연구를 통해 항암, 항말라리아와 항산화 작용 등에 효과가 있는 것으로 보고되었다(Niwat et al., 2015). 명월초의 효능과 관련된 논문은 기존에 다수 보고되었지만 명월초에 감염하는 곰팡이와 세균과 같은 병원체, 특히 바이러스에 대한 연구는 많이 진행되지 않았다. 기존에 명월초에서 보고된 바이러스는 Cucumber mosaic virus (Kwon and Hong, 2017), Broad bean wilt virus 2 (Kwak et al., 2017)가 보고되어 있다.
Broad bean wilt virus (BBWV)는 Comoviridae, fabavirus에 속하는 식물바이러스로 양성 단일 가닥인 분절게놈 RNA1과 RNA2로 구성된다. RNA1에는 5' 말단으로부터 cofactor protease (Co-pro), NTP-binding motif (NTBM), genome-linked viral protein (VPg), protease (Pro)와 RNA-dependent RNA polymerase (RdRp)가 암호화되어 있다. RNA2에는 5' 말단으로부터 movement protein (MP), large coat protein (LCP)과 small coat protein (SCP)이 암호화되어 있다(Koh et al., 2001). 또한 BBWV는 매우 넓은 기주범위를 가지며 Aphis gossypii와 Myzus persicae 등 진딧물에 의해 비영속적으로 전염된다(Lisa and Boccardo, 1996; Stubbs, 1960). 이러한 BBWV 분리주들은 혈청학적 분석과 분자적 특성에 의해 BBWV1과 BBWV2로 구분된다(Kobayashi et al., 1999). 특히 BBWV2의 RNA1과 RNA2는 계통학적 분석을 통해 크게 group I과 II로 구분된다(Seo et al., 2017).
본 연구는 명월초에서 분리된 두 분리주 BBWV2-Gyp와 BBWV2-SN에 대한 생물학적 특성과 유전적 특성을 규명하고자 하였다.
재료 및 방법
시료 채집 및 바이러스 동정
2016년 춘천농업기술원의 온실에서 명월초 상엽에서 바이러스성 모자이크와 황화 증상을 확인하여 채집하였다(Fig. 1). 바이러스 병징을 보이는 잎을 RT-PCR 검정을 진행하기 위해 total RNA를 Phenol-PCI 추출법으로 추출하였다. 바이러스를 확인하기 위한 RT-PCR은 M-MLV Reverse Transcriptase (Promega), i-Taq polymerase (iNtRON)와 fabavirus genus-specific 프라이머(5'-AAATATTAAAACAAACAGCTTTCGTT-3'와 5'-TTCAAAGCTCGTGCC ATNTYATTKGC-3')를 이용해 진행하였다(Ferrer et al., 2007). RT-PCR은 역전사 반응(42°C/60 min) 후 92°C에서 5 min 동안 변성한 뒤, 95°C/30 s, 50°C/30 s, 72°C/1 min 과정을 35회 반복하였고 72°C에서 5 min 동안 반응시켰다. 증폭된 PCR 산물은 Midori green으로 염색한 1% 아가로즈 겔에서 30 min 전기영동 후 UV광 하에서 확인하였다.
기주반응 실험
바이러스의 기주반응을 관찰하기 위해 병징이 보이는 G. procumbens의 잎을 0.01 M 인산 완충용액과 마쇄하고 carborundum과 함께 Chenopodium quinoa, C. amaranticolor, Nicotiana benthamiana, Physalis floridana, Capsicum annuum, Pisum sativum, Phaseolus angularis에 즙액 접종하였다. 바이러스의 감염여부는 BBWV 특이 프라이머(5'-GGTGAGCAGTTTGTCAGAAGT-3'과 5'-CCAGATAATGCATATTCCACC-3')를 사용한 RT-PCR검정과 증식기주인 명아주에서 back inoculation을 통해 확인하였다. 확인된 BBWV2-Gyp의 기주반응은 기존에 보고된 BBWV2-SN과 BBWV2-RP3의 기주반응과 함께 비교하였다.
계통학적 분석
BBWV2-Gyp의 분자적 계통분석을 위해 RNA2 전장 염기서열을 결정하여(Macrogen, Korea), BBWV2-Gyp RNA2의 염기서열을 National Center for Biotechnology Information (NCBI)의 GenBank에 등록(accession No. LC497425)하였다. BBWV2-Gyp 게놈의 계통 분석은 NCBI에서 다양한 BBWV2 분리주들의 염기서열을 수집하여 유전적 상동성을 확인하였다. 또한, 바이러스의 유연관계 분석을 위해 BBWV1-PV132와 BBWV1-Ben을 outgroup으로 사용하였다. 계통학적 분석은 MEGA7 소프트웨어의 Maximum likelihood method (1,000 bootstrap)를 이용하였다.
결과 및 고찰
바이러스성 모자이크와 황화 증상을 보이는 명월초에 감염된 바이러스를 확인하기 위해 명월초 상엽을 N. benthamiana에 즙액 접종하여 바이러스를 증식시킨 후 C. quinoa, C. amaranticolor, N. benthamiana 등에 접종하여 병징을 관찰하였고(Fig. 2), C. quinoa와 C. amaranticolor의 상엽에서 바이러스성 괴사증상이 나타났다. 병징이 발생한 C. quinoa의 잎에서 total RNA를 추출하여 다양한 genus-specific 프라이머를 이용하여 RT-PCR을 수행하였고, fabavirus genus-specific primer를 사용한 반응에서 390 bp의 증폭산물을 확인하였다. 증폭된 RT-PCR산물의 염기서열을 결정하여 분석한 결과 fabavirus에 속하는 Broad bean wilt virus 2 (BBWV2)임을 확인하였고 BBWV2-Gyp로 명명하였다.

Fig. 2.
Symptoms of BBWV2-Gyp on indicator plants. Photographs were taken at 1-2wpi (weeks post-inoculation). (A); upper leaves of C. quinoa, (B); inoculated leaves of C. quinoa, (C); upper leaves of C. amaranticolor, (D); inoculated leaves of C. amaranticolor, (E); upper leaves of N. benthamiana, (F); upper leaves of P. floridana, (G); upper leaves of C. annuum 'Sin-hong', (H); upper leaves of P. angularis, (I); upper leaves of P. sativum.
BBWV2-Gyp의 기주반응을 확인하고자, 가지과, 비름과, 콩과 등 여러 지표식물에 접종하여 병징을 관찰하였다. 또한, 기존에 명월초에서 분리된 BBWV2-SN (Kwak et al., 2017)과 고추에서 분리된 BBWV2-RP3 (Kwak et al., 2013)를 대조군으로 하여 BBWV2-Gyp의 기주반응을 비교 분석하였다. 세 BBWV2 분리주들은 N. benthamiana와 C. quinoa에서 모두 전신감염하는 특성을 보인 반면. BBWV2-Gyp는 C. quinoa에서 상단부 괴사를 유도하는 특이성을 보였다(Table 1, Fig. 2). 특히 명월초 분리주인 BBWV2-Gyp와 BBWV2-SN은 C. amaranticolor와 Phaseolus angularis에서 전신감염하였으나 BBWV2-RP3는 전신감염하지 않는 특성을 보였다. 또한, P. angularis에서 BBWV2- Gyp는 모자이크, BBWV2-SN은 괴사병반 병징을 보였고 C. amaranticolor에서 BBWV2-Gyp는 괴사 병징, BBWV2- SN은 malformation 병징을 보이는 특성을 볼 때 기주에 따라서는 명월초 분리주간 병원성의 차이가 있음을 확인할 수 있었다(Table 1, Fig. 2). 반면에, Capsicum annuum에서 BBWV2-RP3는 전신감염 하였으나, 명월초 분리주들의 경우 감염하지 않는 특성을 보였다. BBWV2-Gyp와 BBWV2-RP3의 경우 Pisum sativum에서 전신감염하지 않았다(Table 1, Fig. 2). 또한, BBWV2-Gyp는 Physalis floridana에서 두 분리주와 달리 전신감염하지 않았다(Table 1, Fig. 2). 위의 결과를 토대로 BBWV2 분리주들의 기주반응을 비교한 결과, 같은 종의 식물에서 분리된 BBWV2-Gyp와 BBWV2-SN은 여러 지표식물에 대한 전신감염 능력이 유사하였으나, 병원성에서는 명확한 차이를 확인할 수 있었다.
Table 1. Host reaction of indicator plants by BBWV2-Gyp
BBWV2-Gyp와 다른 BBWV2 분리주들과의 염기서열을 비교 분석하기 위해 BBWV2 RNA2 전장 프라이머를 사용하여 BBWV2-Gyp RNA2를 증폭한 후 클로닝하여 전장의 염기서열을 결정하였다. BBWV2-Gyp의 RNA2 (GenBank Accession No. LC497425)의 전체 염기서열을 BBWV2 분리주들과 Nucleotide BLAST로 비교 분석하였을 때 90% 이하의 상동성을 보이는 것으로 확인되었다(Table 2). 그러나 명월초 분리주 BBWV2-SN (GenBank Accession No. KX686590)과는 99.0%로 매우 유사한 염기서열 상동성을 보였다. 또한, BBWV2-Gyp와 BBWV2- SN의 movement protein (MP), large coat protein (LCP), small coat protein (SCP)을 아미노산 수준에서 비교 분석한 결과, 염기서열 비교 결과와 마찬가지로 MP, LCP 및 SCP는 각각 98.5%, 98.8%, 98.5%로 높은 상동성을 보였다(Table 2). 추가적으로 기존에 보고된 BBWV2 분리주들과 BBWV2-Gyp의 전장 염기서열과 세 유전자들의 아미노산 서열들을 계통학적으로 분석하였다. 계통학적 분석 결과, BBWV2-Gyp RNA2의 전장 염기서열과 세 유전자들의 아미노산 서열은 모두 BBWV2-SN와 분자계통학적으로 가장 가까운 것으로 확인되었다(Fig. 3). BBWV2의 RNA1과 RNA2는 계통학적 분석을 통해 group I과 II로 구분되어지며 그 중에서도 group II는 II-a, II-b와 II-c로 세분화된다(Kwak et al., 2013). 이를 바탕으로 BBWV2-Gyp 분자적 유연관계를 분석한 결과, BBWV2-Gyp는 BBWV2-SN과 함께 group II로 구분되었으며 II-c에 속하는 것으로 확인되었다(Fig. 3).
Table 2. Sequence homologies (%) of complete genome and three genes (MP, LCP, SCP) between RNA2 of BBWV2-Gyp and other BBWV2 isolates
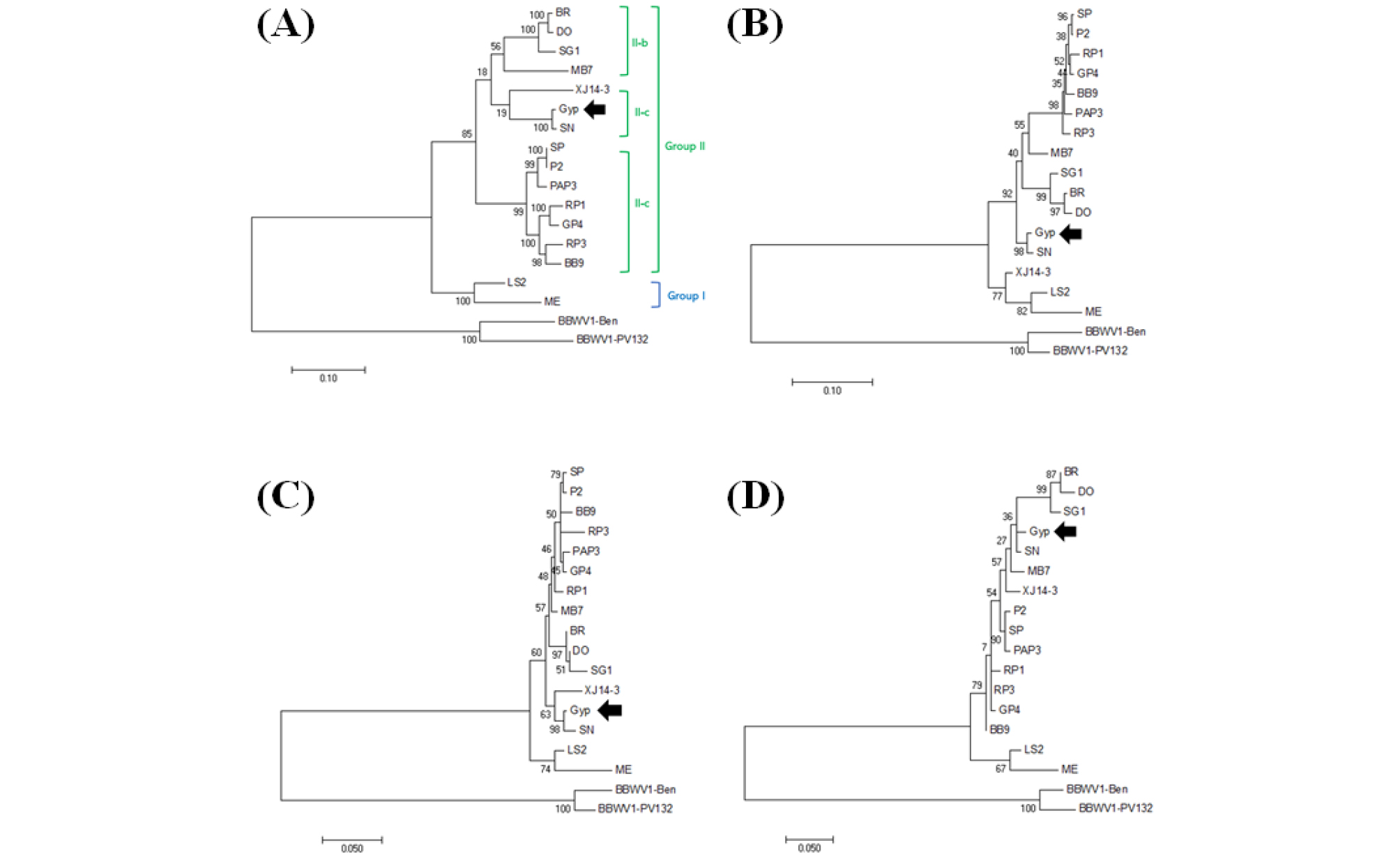
Fig. 3.
Phylogenetic analysis of the complete BBWV2 RNA2 nucleotide sequence (A) and amino acid sequence (MP (B); LCP (C); SCP (D)). The phylogenetic tree was bootstrapped using 1,000 replications by MEGA 7 soft ware. The following sequence of other BBWV2 species were obtained from GenBank database for comparisons. BBWV2-BB9, KC625508; BBWV2-BR, KX234810; BBWV2-DO, KT246496; BBWV2-GP4, KC625510; BBWV2-Gyp, LC497425; BBWV2-LS2, KM076649; BBWV2-MB7, AB013616; BBWV2-ME, AF225954; BBWV2-P2, KC625512; BBWV2-PAP3, KC625515; BBWV2-RP1, KT380023; BBWV2-RP3, JX183226; BBWV2-SG1, KJ789137; BBWV2-SN, KX686590; BBWV2-SP, KC625518; BBWV2- XJ14-3, HQ283389; BBWV1-Ben, KT988974; BBWV1-PV132, NC005290.
두 명월초 분리주간에 매우 높은 유전적 상동성과 기주반응 결과를 토대로 두 명월초 분리주는 명월초에서 특이적으로 적응한 결과로 생각된다. 또한, 기주범위와 병징의 변화에 대한 차이는 이전 연구에서 BBWV2 MP의 424번째 아미노산이 arginine일 때 고추에서 심한 모자이크, 잎의 변형 등의 병징을 유도하고 lysine일 때 약한 모자이크 병징을 유도하는 것으로 확인된 바 있다(Seo et al., 2017). 이를 토대로 BBWV2-Gyp와 BBWV2-SN의 MP 424번째 아미노산을 확인해본 결과, 각각 arginine과 lysine으로 확인되었고 이러한 유전적 변이가 고추를 포함하는 여러 기주에서 병징을 결정하는 결정인자일 것으로 생각되었다. 따라서 명월초에서 분리된 두 BBWV2의 비교 분석 결과는, 기주식물에서의 BBWV2의 감염성과 병징의 결정인자에 대한 연구를 위한 기초자료로 활용 될 것으로 사료된다.
요 약
바이러스성 모자이크와 황화 증상을 보이는 명월초가 춘천에서 채집되었다. 명월초에 감염된 바이러스는 genus- specific 프라이머를 이용한 RT-PCR검정을 통해 BBWV2로 확인되었다. 여러 지표 식물에 대한 기주반응을 비교했을 때 BBWV2-Gyp와 기존에 명월초에서 분리된 BBWV2-SN은 비슷한 기주 범위 패턴을 갖는 것으로 나타났다. 특히 BBWV2-Gyp와 BBWV2-SN은 P. angularis에서 전신감염하지만 각각 모자이크와 괴사병반을 유도하였다. 바이러스의 유전적 특성을 조사하기 위해, BBWV2-Gyp RNA2의 염기서열을 결정하고 다른 BBWV2 분리주들과 비교하였다. 서열 상동성과 계통학적 분석은 BBWV2-Gyp가 BBWV2-SN과 매우 가까운 근연관계를 가지는 것으로 확인되었다. 특히, BBWV2-Gyp RNA2의 유전자들을 BBWV2-SN과 비교했을 때 MP, LCP 및 SCP는 아미노산 수준에서 각각 98.5%, 98.8%, 98.5%의 상동성을 보였다. 두 명월초 분리주들은 높은 염기서열 상동성을 보였으나 구별되는 생물학적 특성을 가졌다.





