서 론
재료 및 방법
호염성 세균 분리 및 배양
16S rDNA 염기서열의 계통학적 분석
세포외 분해 효소 생산능 분석
옥신(Auxin) 생산능 분석
결과 및 고찰
호염성 세균 분리
16S rDNA 염기서열의 계통학적 분석
분해효소 및 옥신 생산능 분석
요 약
서 론
최근 식품산업에서 건강기능성 소재나 건강보조제의 개발을 위한 방법 중 하나로 미생물 발효에 대한 관심이 높아지고 있으며, 이를 이용한 생물학적 전환 공정이 식품산업에서 많이 사용되고 있다(Singh et al., 2016).
발효는 식품이나 식품 원료에 자연적으로 존재하거나 인위적으로 첨가한 미생물을 사용하여 식품이나 식품 원료인 천연물이 가진 성분이나 소재 자체의 기능성이나 활용성을 증진시키는 방법으로 발효 과정 중 미생물들이 생산하는 각종 효소들에 의해 식품 성분에 변화가 생김으로 인해 맛이나 풍미를 증가시키는 것으로 알려져 있으며(Bae et al., 2019), 특히 식품가공 분야에서 미생물 유래 효소의 사용은 제빵, 주스 가공, 곡물 가공, 식품 보존 및 가공, 유지 및 생선 가공, 동물사료 등 다양한 제품의 생산에 protease, amylase, lipase 등을 적용하고 있다(Singh et al., 2016; Mayuri et al., 2019).
또한, 화장품 산업에서도 천연물이나 천연물 유래 추출물 등을 사용하여 미생물 발효를 이용한 천연물이나 추출물 등의 성분 변화를 유도하여 항염 효과나 항산화 활성이 증대되는 기능성 화장품 소재 개발에 미생물 발효가 많이 활용되고 있으며 Bacillus subtilis 균주와 뽕잎을 이용한 발효를 통하여 미생물 균주가 생산하는 단백질 가수분해효소의 작용으로 단백질이 분해되어 저분자 펩타이드의 함량이 증가함으로 인해 아미노산 함량이 증가하고 항산화 활성이 증가하였다는 연구결과도 있다(Seo et al., 2017).
산업계에서 이용되고 있는 효소는 단백질 분해효소(약 60%)와 탄수화물 분해효소(약 28%)가 대부분 사용되고 있으며 건강증진 식품으로 가장 많이 사용되고 있는 홍삼을 이용하여 효소 처리에 의한 추출 수율 향상과 항산화 활성 향상 및 우수한 진세노사이드로 전환을 위하여 amylase와 glucoamylase를 활용한 연구(Kim et al., 2011)와 함께 최근에는 홍삼 추출물의 제조에서 protease를 활용하는 연구도 보고되고 있다(Kim et al., 2019).
일반적으로 호염성 미생물이 생산하는 protease는 수산부산물을 이용한 분야에서도 사용되어 호염성 protease를 이용하여 수산물의 껍질에서 동물성 콜라겐의 가수분해산물을 얻을 수 있기에 수산 폐기물을 활용한 기능성 소재 개발이라는 측면에서도 이러한 protease 또는 protease를 생산하는 미생물을 이용하는 방법이 연구되고 있으며(Wu et al., 2008; Mayuri et al., 2019), 염장식품을 위한 미생물은 고 염농도 뿐만 아니라 산성 또는 염기성 조건에서도 생장이 가능하며 강력한 protease 활성을 지닌 것이 보다 이용 가능성이 높다. 그리고 미생물이 생산하는 protease는 생체 촉매로서 친환경적이고 기질 특이성이 뛰어난 장점을 가지고 있으나 화장품에 배합했을 때 안정성이 떨어져 화장품 원료로 사용하는데 한계를 가지고 있기에 이런 문제를 해결하기 위해 높은 염농도나 산성 또는 염기성 조건에서도 생장이 가능한 미생물 유래의 효소를 이용함으로 해결이 가능할 것으로 생각된다.
최근 비료사용, 화석연료 과다사용, 기후 변화 등 인위적인 인간 활동의 결과로 민물의 염도 변화가 증가하고 있다는 보고(Olson, 2019)가 있었으며, 특히 서천특화시장 개천민물의 경우 주변에 수산시장이 있으며 이는 소금을 많이 사용하는 장소 중 대표적인 곳이 수산시장이기도 하기에 국내 민물시료로 호염균 존재 가능성의 확인을 위하여 본 연구에 사용하였다.
따라서 본 연구에서는 3곳의 국내 민물 시료(순천만 연못물, 서천특화시장 개천민물, 밀양 단장천 민물)에서 호염성 미생물을 분리하고 식품 및 화장품 산업용 효소시장에 응용 가능한 효소 3종(amylase, lipase, protease)의 활성을 중심으로 분리된 미생물의 다양한 분해 효소 생산 특성 탐색과 함께 다양한 미생물에 의해 식물의 세포신장, 발아, 기관의 분화 등에 관여하는 식물호르몬 auxin이 유도 될 수 있다는 것이 보고(Jung et al., 2006; Jung et al., 2007)된 것으로부터 작물의 생육을 촉진하는 기능을 함으로써 화학비료를 대체하는 미생물비료로서의 활용 가능성을 확인하기 위한 auxin 생산능 분석을 실시하였다. 이는 국내 호염성 미생물 자원의 가치를 제고하고 식품 및 화장품 산업용 효소 관련 생물공학 연구와 미생물비료 연구의 기본 생물소재로 활용이 가능할 것으로 예상된다.
재료 및 방법
호염성 세균 분리 및 배양
국내 민물 시료(순천만 연못물, 서천특화시장 개천민물, 밀양 단장천 민물)로부터 호염성 미생물 분리를 위해 각각의 샘플을 멸균된 0.85% 생리식염수에 첨가하여 voltexing으로 현탁하였다. 현탁한 샘플 1ml을 사용하여 10-1-10-4 배로 단계희석 한 후, 일반 증식배지로 해양미생물 전용배지인 marine agar(BD, USA) 배지에 3% NaCl을 첨가한 고체배지를 제작하여 희석액 1ml을 도말하여 37°C에서 호기적으로 호염미생물을 배양하였다. 배양 후 선택적으로 배지상에 나타나는 균의 크기, 모양, 색깔 등 형태학적 모습을 관찰한 후 동일한 고체배지를 사용하여 추가적으로 single colony isolation을 수행하였다. 순수분리된 균주의 혼합배지에서의 생육 가능성을 확인하기 위하여, nutrient agar(BD, USA), R2A agar(BD, USA), 및 tryptic soy agar(BD, USA)에 평판 도말법을 이용하여 37°C에서 7일간 정치 배양을 하였다. 그리고 분리된 균주들이 호염성 균주로써 5%와 10% NaCl 농도에서도 생육이 가능한지를 확인하기 위하여 marine agar 배지에 6%와 9% NaCl을 첨가한 배지를 제작하여 분리 균주들의 생육을 확인하였으며 최적의 생육 pH 조건을 확인하기 위하여 pH를 5, 7, 9로 각각 조절한 marine agar 배지에 분리 균주들의 생육을 확인하였다.
16S rDNA 염기서열의 계통학적 분석
국내 민물 시료로부터 호기적 배양 조건에서 분리된 균주들의 분자생물학적 동정을 위해 marine agar(BD, USA) 배지에 각각 분리된 균주의 colony가 배양된 상태의 고체 배지를 ㈜바이오팩트에 보내어 16S rDNA 염기서열의 분석을 의뢰하였으며 분석된 16S rDNA 염기서열로부터 가장 유사한 근연 균주의 확인을 위하여 ㈜천랩의 웹 기반 분석 데이터베이스인 EzBioCloud에 접속하여 16S-based ID 분석 앱(https://www.ezbiocloud.net/)을 사용하였다. 계통학적 분석은 BioEdit과 MEGA X 프로그램을 이용하여 확인하였다.
세포외 분해 효소 생산능 분석
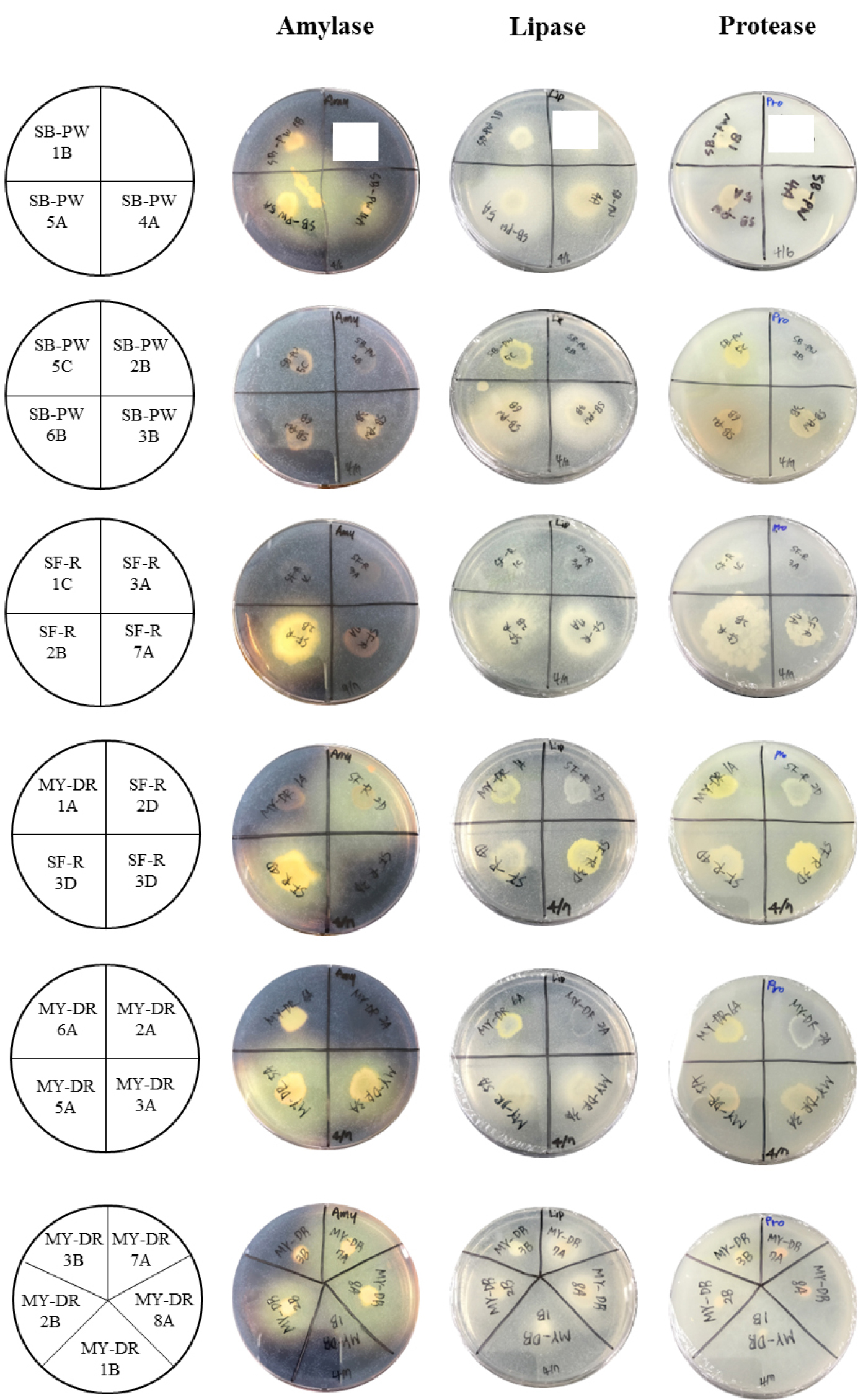
분리된 호염성 미생물의 세포외 분해 효소 amylase, lipase, protease 생산능 확인을 위하여 각각의 효소와 특이적으로 반응할 기질 성분이 포함된 고체평판 선별배지를 사용하였다. 먼저 amylase 생산능은 0.2% soluble starch(BD, USA)를, lipase 생산능은 1% Tween 80(Sigma, USA)을, protease 생산능은 2% skim milk(BD, USA)를 기질로 선택하여 marine agar(BD, USA) 배지에 각각 첨가하여 제조하였으며 분리된 균주를 직접 접종하여 37°C에서 7일 배양한 후 투명환(Clear zone)의 직경으로 조사하였다. 분리된 균주의 효소활성 분해능 평가는 배양 후 나타나는 접종균 주위의 투명환의 크기(+++ : > 7mm, ++ : 4-6mm, + : 1-3mm)로 나타내었다.
옥신(Auxin) 생산능 분석
분리된 균주의 auxin 생산능은 0.1% L-tryptophan이 첨가된 Marin broth(BD, USA) 배지에 순수분리된 colony를 tooth-picking 한 후 37°C에서 5일 배양하여 Salkowski 시약(35% HClO4 50ml + 0.5 M FeCl3 1ml) 800µl을 배양 상등액 400µl 에 섞어준 후 어두운 곳에서 30min 반응시켜 육안으로 확인하여 붉은 색깔 변화에 따라 옥신 생성능 정도를 표시하였다(붉은색: +; 무색: -).
결과 및 고찰
호염성 세균 분리
국내 민물 시료(순천만 연못물, 서천특화시장 개천민물, 밀양 단장천 민물)로부터 식품과 화장품 산업에의 활용이 용이할 것으로 생각되는 호기적으로 생육 가능한 호염성 미생물을 분리하고자 marine agar 배지에 시료를 희석, 도말하여 배양한 후, 배양된 colony의 모양, 색깔 등 형태학적 특징을 육안으로 구분이 가능한 균주들을 대상으로 동일한 고체배지를 사용하여 2차로 단일 균주 분리를 수행하였다. 그 결과 Table 1에서 나타낸 것처럼 순천만 연못물 시료로부터 7균주, 서천특화시장 개천 민물 시료에서 7균주, 밀양 단장천 민물 시료로부터 10균주가 각각 분리되었으며 본 실험을 통하여 호염성 미생물 총 24균주를 순수 분리하였다. 또한 주요성분이 무기염으로 이루어진 marine agar 배지는 해양미생물 배양에 유리한 배지이기에 산업적 활용가능성을 확인하기 위하여 대량 배양 등에 많이 활용되는 혼합 배지(nutrient agar, R2A agar, tryptic soy agar)에서의 분리 균주들의 생육 가능성을 확인한 결과 24균주 중 1균주(No. 8)를 제외한 23균주(95.8%)가 최소 1종류 이상의 혼합 배지에서 생육이 가능한 것을 확인하였다. 그리고 분리 균주가 높은 염농에서도 생육이 가능한지를 확인하기 위하여 marine agar 배지에 6%와 9% NaCl을 각각 첨가한 배지를 제작하여 분리 균주들의 생육을 확인한 결과, 6% 와 9% NaCl 조건에서는 13균주의 생육이 가능한 것을 확인하였다. 또한 최적의 생육 pH 조건을 확인하기 위하여 pH를 5, 7, 9로 각각 조절한 marine agar 배지에 분리 균주들의 생육을 확인해본 결과 분리된 모든 균주가 pH7에서 생육이 가능하였으며 이중 3균주(No. 20, 23, 24)만이 pH5와 pH9에서 생육 가능한 것으로 확인되었다(Table 1).
Table 1.
Isolation and identification of halophilic microorganisms isolated from freshwater samples in Korea
| No. | Source | Isolate number | NA# | R2A | TSA | MA* | ||||
| NaCl (6%) | NaCl (9%) | pH5 | pH7 | pH9 | ||||||
| 1 | Suncheonman pond | SB-PW 4A | +a | + | + | -b | - | - | + | - |
| 2 | SB-PW 5A | + | + | + | - | - | - | + | - | |
| 3 | SB-PW 1B | + | + | + | + | + | - | + | - | |
| 4 | SB-PW 2B | + | - | + | + | + | - | + | - | |
| 5 | SB-PW 3B | + | + | + | - | - | - | + | - | |
| 6 | SB-PW 6B | + | + | + | - | - | - | + | - | |
| 7 | SB-PW 5C | + | + | + | - | - | - | + | - | |
| 8 | Seocheon Special Market stream | SF-R 3A | - | - | - | + | + | - | + | - |
| 9 | SF-R 7A | + | + | + | + | + | - | + | - | |
| 10 | SF-R 2B | + | + | + | + | + | - | + | - | |
| 11 | SF-R 1C | - | + | - | - | - | - | + | - | |
| 12 | SF-R 2D | + | + | + | + | + | - | + | - | |
| 13 | SF-R 3D | + | + | + | + | + | - | + | - | |
| 14 | SF-R 4D | - | + | + | + | + | - | + | - | |
| 15 | Miryang Danjangcheon | MY-DR 1A | + | + | + | + | + | - | + | - |
| 16 | MY-DR 2A | + | + | + | + | + | - | + | - | |
| 17 | MY-DR 3A | + | + | + | - | - | - | + | - | |
| 18 | MY-DR 5A | + | + | + | - | - | - | + | - | |
| 19 | MY-DR 6A | + | + | + | + | + | - | + | - | |
| 20 | MY-DR 7A | + | + | + | + | + | + | + | - | |
| 21 | MY-DR 8A | + | + | + | - | - | - | + | - | |
| 22 | MY-DR 1B | + | + | + | - | - | - | + | - | |
| 23 | MY-DR 2B | + | + | + | - | - | + | + | + | |
| 24 | MY-DR 3B | + | + | + | + | + | + | + | + | |
16S rDNA 염기서열의 계통학적 분석
국내 민물 시료(순천만 연못물, 서천특화시장 개천민물, 밀양 단장천 민물)로부터 호기적으로 분리된 27균주의 16S rDNA 염기서열을 바탕으로 EzBioCloud의 16S-based ID 분석서비스를 사용하여 미생물 동정을 실시한 결과 크게 3문 3강 9목 11과 12속 17종으로 나타났으며(Table 2), 분리 균주와 근연 균주 및 상동성을 Table 3에 나타내었다. 또한 분리 동정된 균주들 간의 유연관계를 확인하기 위하여 계통수를 작성하였다(Fig. 1).
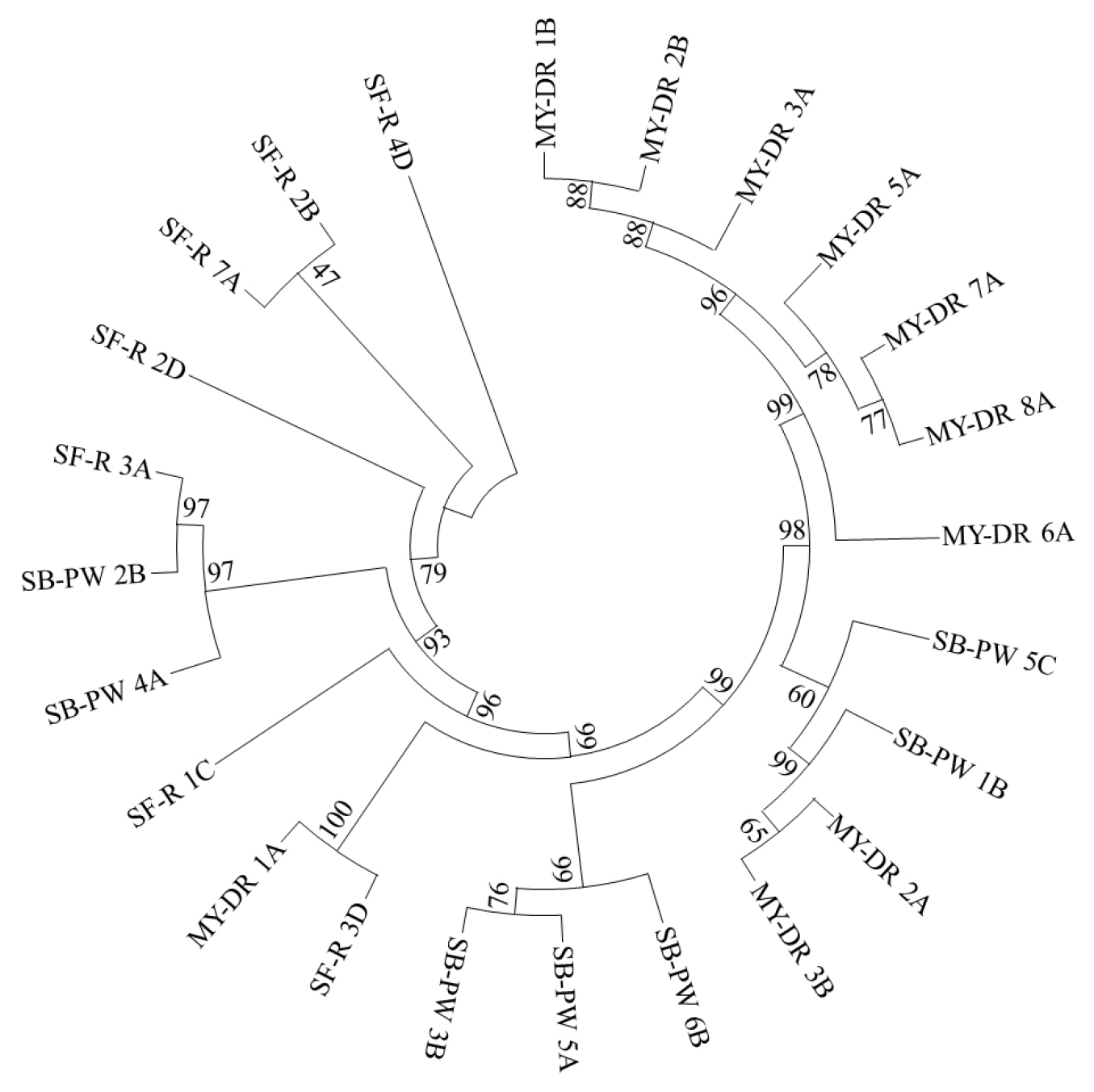
Fig. 1.
Evolutionary relationships of taxa based on the 16S rDNA sequences of freshwater bacteria in Korea. The evolutionary history was inferred using the neighbor-joining method (Saitou and Nei, 1987). The bootstrap consensus tree inferred from 1000 replicates represented the evolutionary history of the analyzed taxa (Felsenstein, 1985). Branches corresponding to partitions reproduced in less than 50% bootstrap replicates were collapsed. The evolutionary distances were computed using the Maximum Composite Likelihood method (Tamura et al., 2004). The distances are expressed as the number of base substitutions per site. This analysis involved 24 nucleotide sequences. Codon positions included were 1st+2nd+3rd+Noncoding. All ambiguous positions were removed for each sequence pair (pairwise deletion option). There were a total of 1571 positions in the final dataset. Evolutionary analyses were conducted in MEGA X (Kumar et al., 2018).
Table 2에서 보는 바와 같이 Proteobacteria(Gammaproteobacteria)가 58.4%로 가장 우점도가 높았고, Firmicutes (Bacilli)가 33.3%, Actinobacteria(Actinomycetia)가 8.3%로 나타났다. 가장 우점도가 높은 총 14균주가 분리된 Proteobacteria 문에서는 Gammaproteobacteria 강으로 분류되는 균주만 분리되었으며 Aeromonadaceae 42.9%, Lysobacteraceae와 Moraxellaceae가 각각 21.4%, 그리고 Enterobacteriaceae와 Pseudomonadaceae가 각각 7.15%로 구성되어 있는 것을 확인하였으며 총 5과로 5속 9종이 분리되었다. Firmicutes 문은 Bacillaceae와 Carnobacteriaceae가 각각 37.5%, Paenibacillaceae와 Staphylococcaceae가 각각 12.5%로 구성되었으며, 총 4과로 5속 6종이 분리되었다. 마지막으로 2균주가 분리된 Actinobacteria 문에서는 Microbacteriaceae와 Micrococcaceae가 각각 50%로, 2과로 2속 2종이 분리되었다.
Table 2.
Phylum analysis of halophilic microorganisms isolated from freshwater samples in Korea
분해효소 및 옥신 생산능 분석
분리된 호염성 미생물 균주들의 식품 및 화장품 산업에의 응용가능성을 탐색하기 위하여 분해효소 생산능의 확인을 실시하였다(Fig. 2). 또한, 일반적으로 토양 미생물들은 토양으로 유입되는 낙엽, 볏짚 등 유기물들을 세포외 분해효소를 분비하여 영양물질 등으로 분해하기 때문에 호르몬(hormone), 비타민(vitamin), 식물생장 촉진물질 등을 생산하여 식물의 생장을 돕는다고 알려져 있기에 미생물 비료로서의 산업적 응용 가능성을 확인하기 위하여 옥신 생산능 분석을 실시하였다.
Table 3에 나타낸 것처럼 분리된 24균주에서 7균주를 제외한 모든 균주에서 한 가지 이상의 분해 효소 활성이 존재하는 것을 확인하였으며, 두 가지 이상의 효소 활성을 가지는 균주도 11균주(기탁번호: NMC4-B260, NMC4-B261, NMC4-B262, NMC4-B264, NMC4-B265, NMC4-B271, NMC4-B273, NMC4-B276, NMC4-B277, NMC4-B279, NMC4-B280)도 확인되었다. 또한, 옥신 생산능을 확인한 결과, 3균주(기탁번호: NMC4-B272, NMC4-B277, NMC4-B278)에서 생산능이 확인되었으며 서천특화시장 개천 민물 시료에서 1균주, 밀양 단장천 민물 시료에서 2균주가 분리되었으며 순천만 연못물 시료에서는 옥신 생산능을 가진 균주는 분리되지 않았다. 이는 국내 민물 시료가 식물 생장을 촉진시킬 수 있는 미생물비료 연구에 활용할 수 있는 미생물 탐색 소재로써 중요한 원천소재로서의 활용가치가 높을 것으로 예상되는 결과이기도 하다. 본 연구에서 수행한 결과들은 국내 미생물 생물 자원의 다양성 확보 차원에서 큰 의미를 찾을 수 있을 것이며 국내 민물 시료에서 분해 효소 탐색을 위한 새로운 균주 탐색 가능성과 식품과 화장품 산업에 적용을 위한 효소 관련 생물공학 및 미생물비료 연구의 기본 생물소재로 활용이 가능할 것으로 예상된다. 또한 본 연구를 통하여 분리한 모든 균주들은 한국생명공학연구원 미생물가치제고사업단에 기탁하였다.
Table 3.
Representative sequences of halophilic microorganisms isolated from freshwater samples in Korea
| No |
Isolate Name | Top hit taxon | Top hit strain | Similarity (%) |
Extracellular enzyme activity | Auxin |
Deposited number | ||
| Amylase | Lipase | Protease | |||||||
| 1 | SB-PW 4A | Desemzia incerta | DSM 20581 | 99.86 | +++b | +++ | -a | - | NMC4-B260 |
| 2 | SB-PW 5A | Stenotrophomonas pavanii | DSM 25135 | 100 | +++ | +++ | - | - | NMC4-B261 |
| 3 | SB-PW 1B | Acinetobacter radioresistens | DSM 6976 | 100 | +b | +++ | - | - | NMC4-B262 |
| 4 | SB-PW 2B | Desemzia incerta | DSM 20581 | 99.86 | - | - | - | - | NMC4-B263 |
| 5 | SB-PW 3B | Stenotrophomonas pavanii | DSM 25135 | 100 | - | +++ | +++ | - | NMC4-B264 |
| 6 | SB-PW 6B | Stenotrophomonas maltophilia | MTCC 434 | 100 | - | +++ | +++ | - | NMC4-B265 |
| 7 | SB-PW 5C | Pseudomonas parafulva | NBRC 16636 | 100 | - | - | - | - | NMC4-B266 |
| 8 | SF-R 3A | Desemzia incerta | DSM 20581 | 99.86 | - | - | - | - | NMC4-B267 |
| 9 | SF-R 7A | Bacillus altitudinis | 41KF2b | 100 | - | - | +++ | - | NMC4-B268 |
| 10 | SF-R 2B | Bacillus licheniformis | ATCC 14580 | 99.86 | - | - | +++ | - | NMC4-B269 |
| 11 | SF-R 1C | Brevibacillus limnophilus | DSM 6472 | 100 | +++ | - | - | - | NMC4-B270 |
| 12 | SF-R 2D | Staphylococcus epidermidis | NCTC 11047 | 100 | +++ | - | +++ | - | NMC4-B271 |
| 13 | SF-R 3D | Micrococcus luteus | NCTC 2665 | 100 | - | - | +++ | + | NMC4-B272 |
| 14 | SF-R 4D | Bacillus idriensis | SMC 4352-2 | 100 | +++ | - | +++ | - | NMC4-B273 |
| 15 | MY-DR 1A | Microbacterium hydrocarbonoxydans | NBRC 103074 | 100 | - | - | - | - | NMC4-B274 |
| 16 | MY-DR 2A | Acinetobacter pittii | CIP 70.29 | 100 | - | - | ++b | - | NMC4-B275 |
| 17 | MY-DR 3A | Aeromonas media | CECT 4232 | 100 | +++ | +++ | - | - | NMC4-B276 |
| 18 | MY-DR 5A | Aeromonas hydrophila subsp. hydrophila | ATCC 7966 | 100 | +++ | +++ | - | + | NMC4-B277 |
| 19 | MY-DR 6A | AXOM_s | S611 | 100 | - | - | - | + | NMC4-B278 |
| 20 | MY-DR 7A | Aeromonas hydrophila subsp. hydrophila | ATCC 7966 | 100 | +++ | +++ | - | - | NMC4-B279 |
| 21 | MY-DR 8A | Aeromonas hydrophila subsp. hydrophila | ATCC 7966 | 100 | +++ | +++ | - | - | NMC4-B280 |
| 22 | MY-DR 1B | Aeromonas media | CECT 4232 | 100 | - | - | - | - | NMC4-B281 |
| 23 | MY-DR 2B | Aeromonas media | CECT 4232 | 100 | +++ | - | - | - | NMC4-B282 |
| 24 | MY-DR 3B | Acinetobacter tandoii | DSM 14970 | 99.45 | - | - | - | - | NMC4-B283 |
요 약
본 연구는 국내 민물 시료로부터 분리한 호염성 미생물들의 특성에 관하여 조사하였다. 호염성 미생물의 순수 분리를 위하여 marine agar 배지를 사용하였으며 37°C 에서 호기적으로 배양하였다. 순수 분리 후, 24균주를 분리하였으며 16S rRNA 염기서열 분석 결과를 바탕으로 계통학적 분석을 EzBioCloud 데이터베이스의 16S-based ID 분석 프로그램으로 실시한 결과, 3문, 11과, 12속, 17종으로 구성되어 있는 것을 확인하였다. 그리고 분리한 균주들이 amylase, lipase, protease 같은 산업적으로 유용한 효소를 생산하는지 확인하기 위하여 효소 활성 평가를 실시하였으며, 17균주가 최소 한 종류 이상의 효소 활성을 가지고 있는 것을 확인하였으며 두 가지 효소에 대하여 활성을 가지고 있는 균주도 11균주가 확인되었으며 auxin 생산능을 가진 균주도 3균주가 확인되었다. 이는 본 연구를 통하여 분리한 미생물들의 식품 및 화장품 산업에서의 활용 가능성을 나타내었다. 그러므로 이번 연구는 국내 유전자원 확보 및 가수분해 효소 생산능 개선을 위한 균주 개량에 본 연구를 통하여 순수분리된 균주의 활용이 도움이 될 것으로 생각된다.